Article publié dans Conservation Biology
Prédiction de la diversité génétique des populations naturelles à l’aide de modèles de connectivité des habitats
 Dans un contexte de changements globaux, le maintien d’une espèce peut reposer sur le déplacement des populations dans d’autres zones géographiques afin d’y retrouver des conditions environnementales initiales. Une alternative est offerte par l’adaptation aux nouvelles conditions environnementales locales, sous réserve que les populations renferment une diversité génétique suffisante, cette dernière étant en partie conditionnée par le brassage génétique entre populations. Par conséquent, la connectivité des habitats est devenue une notion clé dans les politiques de conservation puisqu’elle détermine les patrons de dispersion des organismes. En confrontant les résultats de modélisations de réseaux écologiques à des données génétiques issues d’échantillons collectés sur le terrain, deux doctorants de Biogéosciences (Alexandrine Daniel et Paul Savary) valident empiriquement une méthode de modélisation de la connectivité des habitats couramment employée pour l’aménagement du territoire. Leur travail a été publié le 20 janvier 2023 dans la revue Conservation Biology.
Dans un contexte de changements globaux, le maintien d’une espèce peut reposer sur le déplacement des populations dans d’autres zones géographiques afin d’y retrouver des conditions environnementales initiales. Une alternative est offerte par l’adaptation aux nouvelles conditions environnementales locales, sous réserve que les populations renferment une diversité génétique suffisante, cette dernière étant en partie conditionnée par le brassage génétique entre populations. Par conséquent, la connectivité des habitats est devenue une notion clé dans les politiques de conservation puisqu’elle détermine les patrons de dispersion des organismes. En confrontant les résultats de modélisations de réseaux écologiques à des données génétiques issues d’échantillons collectés sur le terrain, deux doctorants de Biogéosciences (Alexandrine Daniel et Paul Savary) valident empiriquement une méthode de modélisation de la connectivité des habitats couramment employée pour l’aménagement du territoire. Leur travail a été publié le 20 janvier 2023 dans la revue Conservation Biology.
La dispersion est un processus écologique majeur, dans la mesure où elle conditionne la démographie et la diversité génétique des populations (et donc leur persistance), la composition des communautés ou encore la transmission d’agents pathogènes. Elle est déterminée en partie par la connectivité des habitats, qui dépend de la structure des paysages (composition et configuration des éléments de la matrice paysagère) et de la capacité des individus à s’y déplacer. Évaluer la connectivité des habitats consiste alors à cartographier les taches d’habitat ainsi que les principaux flux de dispersion d’individus, en s’appuyant sur des hypothèses biologiques définissant un « modèle de connectivité des habitats ». Cette évaluation est importante, tant pour comprendre les processus éco-évolutifs sous-jacents à la dynamique de la biodiversité que pour la mise en œuvre des politiques de conservation de la biodiversité (par exemple TVB et SRADD en France).
Une des méthodes de modélisation couramment utilisées repose sur l’utilisation de graphes paysagers. Elle consiste à représenter le réseau écologique sous la forme d’un graphe, c’est-à-dire d’un ensemble de nœuds représentant les taches d’habitat, reliés par des liens associés à des distances écologiques et reflétant la difficulté de déplacement des individus. Cette représentation, qui permet à la fois d’identifier les principaux corridors de dispersion et les taches d’habitat clés du réseau, présente un fort intérêt opérationnel. Cependant, cet outil de modélisation manquait jusqu’alors d’une validation empirique, notamment concernant le sens biologique des distances écologiques et des indicateurs de connectivité produits. Des analyses récemment publiées dans Conservation Biology se sont appuyées sur des données génétiques (reflétant des flux de gènes, eux même liés aux évènements de dispersion) et les ont confrontées aux graphes paysagers. Elles révèlent que la diversité génétique et le degré de similarité génétique des populations de Paruline caféiette (Setophaga plumbea) étudiées en Guadeloupe sont étroitement corrélées aux propriétés des tâches d’habitat forestier (i.e. nœuds du graphe) qu’elles occupent et à la connectivité entre ces tâches (i.e. les liens qui relient les nœuds du graphe). Ces analyses confirment des résultats précédemment obtenus sur ce territoire démontrant l’influence de la structure du paysage sur les flux de gènes (Khimoun et al., 2017), et valident une méthode de modélisation de la connectivité. Ces analyses révèlent également que le niveau de validation de cette méthode diffère selon la façon dont les graphes sont créés, fournissant ainsi de précieuses informations pour les utilisations futures de ces graphes paysagers à des fin d’aménagement du territoire et de conservation de la biodiversité.
Cette étude s’inscrit dans le cadre du projet CANON (Stéphane Garnier), qui regroupe les laboratoires Biogéosciences, ThéMa et l’Institut de Mathématiques de Bourgogne, et qui vise à étudier les effets de la connectivité paysagère sur la biodiversité dans la région Caraïbes. Les graphes paysagers analysés dans cette étude ont été créés avec le logiciel Graphab (Foltête et al., 2021), dans le cadre d’un partenariat avec le laboratoire ThéMA, qui développe cet outil depuis 2012 et forme des professionnels de la conservation à son utilisation.
Contact chercheur
Stéphane Garnier, UMR Biogéosciences, maître de conférences en écologie, stephane.garnier@u-bourgogne.fr
Correspondant communication du laboratoire
Alexandre Pohl, Biogéosciences, UMR 6282 CNRS, Université de Bourgogne – alexandre.pohl@u-bourgogne.fr
Références
Daniel, A., Savary, P., Foltête, J.-C., Khimoun, A., Faivre, B., Ollivier, A., Éraud, C., Moal, H., Vuidel, G., & Garnier, S. (2022). Validating graph-based connectivity models with independent presence-absence and genetic data sets. Conservation Biology. https://doi.org/10.1111/cobi.14047
Foltête, J. C., Vuidel, G., Savary, P., Clauzel, C., Sahraoui, Y., Girardet, X., & Bourgeois, M. (2021). Graphab: an application for modeling and managing ecological habitat networks. Software Impacts, 8, 100065. https://doi.org/10.1016/j.simpa.2021.100065
Khimoun, Aurélie, William Peterman, Cyril Eraud, Bruno Faivre, Nicolas Navarro, and Stéphane Garnier. 2017. “Landscape Genetic Analyses Reveal Fine-Scale Effects of Forest Fragmentation in an Insular Tropical Bird.” Molecular Ecology 26 (19): 4906–19. https://doi.org/10.1111/mec.14233.
Légende de la figure illustrative
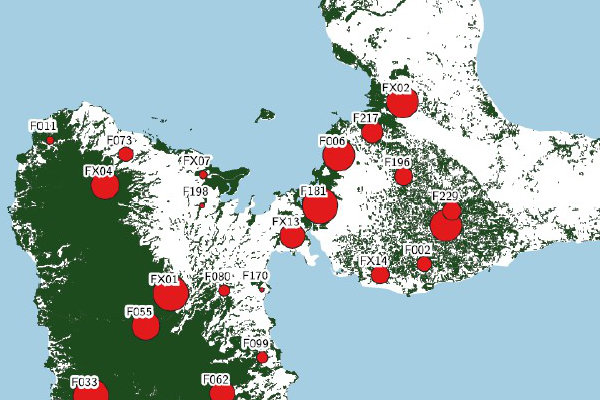
Confrontation d’un graphe de connectivité de l’habitat forestier (figure A) pour la Paruline Caféiette (passereau forestier endémique de la région Caraïbes), avec une représentation spatiale de la diversité génétique des populations échantillonnées chez cette espèce (figure B).
A – Carte représentant les nœuds et des liens du graphe de l’habitat forestier, ainsi que les populations échantillonnées.
B – Carte représentant la diversité génétique spatialisée (richesse allélique) des populations échantillonnées.
- extrait:
- lien_externe:
- kc_data:
- a:8:{i:0;s:0:"";s:4:"mode";s:0:"";s:3:"css";s:0:"";s:9:"max_width";s:0:"";s:7:"classes";s:0:"";s:9:"thumbnail";s:0:"";s:9:"collapsed";s:0:"";s:9:"optimized";s:0:"";}
- kc_raw_content:
Prédiction de la diversité génétique des populations naturelles à l'aide de modèles de connectivité des habitats
 Dans un contexte de changements globaux, le maintien d'une espèce peut reposer sur le déplacement des populations dans d'autres zones géographiques afin d'y retrouver des conditions environnementales initiales. Une alternative est offerte par l'adaptation aux nouvelles conditions environnementales locales, sous réserve que les populations renferment une diversité génétique suffisante, cette dernière étant en partie conditionnée par le brassage génétique entre populations. Par conséquent, la connectivité des habitats est devenue une notion clé dans les politiques de conservation puisqu'elle détermine les patrons de dispersion des organismes. En confrontant les résultats de modélisations de réseaux écologiques à des données génétiques issues d’échantillons collectés sur le terrain, deux doctorants de Biogéosciences (Alexandrine Daniel et Paul Savary) valident empiriquement une méthode de modélisation de la connectivité des habitats couramment employée pour l'aménagement du territoire. Leur travail a été publié le 20 janvier 2023 dans la revue Conservation Biology.
Dans un contexte de changements globaux, le maintien d'une espèce peut reposer sur le déplacement des populations dans d'autres zones géographiques afin d'y retrouver des conditions environnementales initiales. Une alternative est offerte par l'adaptation aux nouvelles conditions environnementales locales, sous réserve que les populations renferment une diversité génétique suffisante, cette dernière étant en partie conditionnée par le brassage génétique entre populations. Par conséquent, la connectivité des habitats est devenue une notion clé dans les politiques de conservation puisqu'elle détermine les patrons de dispersion des organismes. En confrontant les résultats de modélisations de réseaux écologiques à des données génétiques issues d’échantillons collectés sur le terrain, deux doctorants de Biogéosciences (Alexandrine Daniel et Paul Savary) valident empiriquement une méthode de modélisation de la connectivité des habitats couramment employée pour l'aménagement du territoire. Leur travail a été publié le 20 janvier 2023 dans la revue Conservation Biology.La dispersion est un processus écologique majeur, dans la mesure où elle conditionne la démographie et la diversité génétique des populations (et donc leur persistance), la composition des communautés ou encore la transmission d'agents pathogènes. Elle est déterminée en partie par la connectivité des habitats, qui dépend de la structure des paysages (composition et configuration des éléments de la matrice paysagère) et de la capacité des individus à s’y déplacer. Évaluer la connectivité des habitats consiste alors à cartographier les taches d’habitat ainsi que les principaux flux de dispersion d’individus, en s’appuyant sur des hypothèses biologiques définissant un « modèle de connectivité des habitats ». Cette évaluation est importante, tant pour comprendre les processus éco-évolutifs sous-jacents à la dynamique de la biodiversité que pour la mise en œuvre des politiques de conservation de la biodiversité (par exemple TVB et SRADD en France).
Une des méthodes de modélisation couramment utilisées repose sur l’utilisation de graphes paysagers. Elle consiste à représenter le réseau écologique sous la forme d’un graphe, c’est-à-dire d’un ensemble de nœuds représentant les taches d’habitat, reliés par des liens associés à des distances écologiques et reflétant la difficulté de déplacement des individus. Cette représentation, qui permet à la fois d’identifier les principaux corridors de dispersion et les taches d’habitat clés du réseau, présente un fort intérêt opérationnel. Cependant, cet outil de modélisation manquait jusqu’alors d’une validation empirique, notamment concernant le sens biologique des distances écologiques et des indicateurs de connectivité produits. Des analyses récemment publiées dans Conservation Biology se sont appuyées sur des données génétiques (reflétant des flux de gènes, eux même liés aux évènements de dispersion) et les ont confrontées aux graphes paysagers. Elles révèlent que la diversité génétique et le degré de similarité génétique des populations de Paruline caféiette (Setophaga plumbea) étudiées en Guadeloupe sont étroitement corrélées aux propriétés des tâches d’habitat forestier (i.e. nœuds du graphe) qu’elles occupent et à la connectivité entre ces tâches (i.e. les liens qui relient les nœuds du graphe). Ces analyses confirment des résultats précédemment obtenus sur ce territoire démontrant l’influence de la structure du paysage sur les flux de gènes (Khimoun et al., 2017), et valident une méthode de modélisation de la connectivité. Ces analyses révèlent également que le niveau de validation de cette méthode diffère selon la façon dont les graphes sont créés, fournissant ainsi de précieuses informations pour les utilisations futures de ces graphes paysagers à des fin d’aménagement du territoire et de conservation de la biodiversité.
Cette étude s’inscrit dans le cadre du projet CANON (Stéphane Garnier), qui regroupe les laboratoires Biogéosciences, ThéMa et l'Institut de Mathématiques de Bourgogne, et qui vise à étudier les effets de la connectivité paysagère sur la biodiversité dans la région Caraïbes. Les graphes paysagers analysés dans cette étude ont été créés avec le logiciel Graphab (Foltête et al., 2021), dans le cadre d’un partenariat avec le laboratoire ThéMA, qui développe cet outil depuis 2012 et forme des professionnels de la conservation à son utilisation.
Contact chercheur
Stéphane Garnier, UMR Biogéosciences, maître de conférences en écologie, stephane.garnier@u-bourgogne.fr
Correspondant communication du laboratoire
Alexandre Pohl, Biogéosciences, UMR 6282 CNRS, Université de Bourgogne – alexandre.pohl@u-bourgogne.fr
Références
Daniel, A., Savary, P., Foltête, J.-C., Khimoun, A., Faivre, B., Ollivier, A., Éraud, C., Moal, H., Vuidel, G., & Garnier, S. (2022). Validating graph-based connectivity models with independent presence-absence and genetic data sets. Conservation Biology. https://doi.org/10.1111/cobi.14047
Foltête, J. C., Vuidel, G., Savary, P., Clauzel, C., Sahraoui, Y., Girardet, X., & Bourgeois, M. (2021). Graphab: an application for modeling and managing ecological habitat networks. Software Impacts, 8, 100065. https://doi.org/10.1016/j.simpa.2021.100065
Khimoun, Aurélie, William Peterman, Cyril Eraud, Bruno Faivre, Nicolas Navarro, and Stéphane Garnier. 2017. “Landscape Genetic Analyses Reveal Fine-Scale Effects of Forest Fragmentation in an Insular Tropical Bird.” Molecular Ecology 26 (19): 4906–19. https://doi.org/10.1111/mec.14233.
Légende de la figure illustrative
Confrontation d’un graphe de connectivité de l’habitat forestier (figure A) pour la Paruline Caféiette (passereau forestier endémique de la région Caraïbes), avec une représentation spatiale de la diversité génétique des populations échantillonnées chez cette espèce (figure B).
A – Carte représentant les nœuds et des liens du graphe de l’habitat forestier, ainsi que les populations échantillonnées.
B – Carte représentant la diversité génétique spatialisée (richesse allélique) des populations échantillonnées.
